数千万种仍未获知或被误解的病毒可引发疾病(包括新的大流行病),并影响宝贵的陆地和海洋环境的健康。
现在,由弗林德斯大学领导的国际合作小组开发了一条新途径,用于研究“病毒圈”,即世界上所有病毒和噬菌体(攻击细菌的病毒)的基因组。
在GigaScience杂志描述的项目中,弗林德斯微生物组探索加速器(FAME)的研究人员与华盛顿大学教授斯科特·汉德利及其同事一起分析人类群体和珊瑚礁中的病毒,以建立一种识别所有动物、植物和环境中病毒的强大方法,并展示该流程对未来研究的灵活性。
在FAME主任——微生物学家罗伯特·爱德华兹教授和海洋微生物生物学家伊丽莎白·丁斯代尔教授的协助下,该项目对英国大型社区粪便样本中发现的病毒进行了深度宏基因组测试。该研究利用英国的大型数据库调查了数据中的异常情况——包括不同家庭之间病毒基因组的差异。
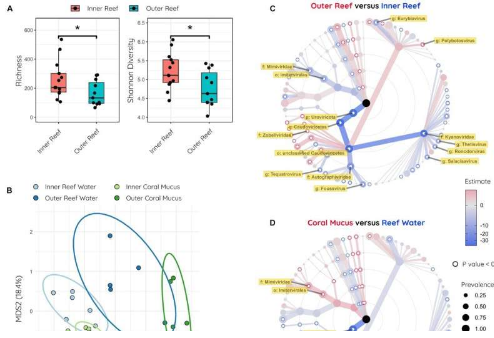
研究人员随后通过研究来自完全不同的环境(从百慕大珊瑚礁中提取的粘液涂层)的一系列病毒数据来测试该方法的可处理性。
“病毒是迄今为止地球上最多样化的生物,但病毒测序的准确识别和注释仍处于早期阶段,”生物信息学和人类微生物学教授爱德华兹说。
“这条新方法试图排除或‘牺牲’数千个基因组序列,以消除异常,并找到每个病毒组特有的新型病毒,以找到疾病的‘特征’。”
该项目被命名为“Hecatomb”,取自古希腊献祭或屠宰动物的传统。
爱德华兹说:“现在我们有了一个新的病毒宏基因组学软件平台,可以在序列大海中找到病毒,以便更好地了解这些新病毒或现有病毒的行为。”
Hecatomb流程和实现。Hecatomb流程分为4个模块。图片来源:GigaScience(2024)。DOI:10.1093/gigascience/giae020
华盛顿大学医学院病理学和免疫学教授Handley博士表示,Hecatomb项目提供了研究与炎症性肠病等疾病相关的病毒和噬菌体的机会。
Handley教授说:“我参与的项目致力于加深对微生物和病毒生态学以及侵入性病原体如何运作并影响人类健康的理解。”
“为了做到这一点,我们需要制作百屠流程来利用高通量测序技术,并集成能够进行病毒群落分析的计算工具。
“我们使用百尸屠杀管道来分类和测试病毒群落成员和功能的改变如何导致疾病。”
第一作者、南澳大利亚生物信息学家MichaelRoach博士表示,该项目发现了许多独特的病毒,为研究病毒如何影响或加剧肠易激疾病等常见疾病提供了一个强有力的起点,甚至可以通过减少白化或污染的影响来拯救珊瑚礁。
“通过我们在宏基因组样本中研究的病毒管道,我们现在可以寻找能够解释疾病状态的病毒或噬菌体特征,包括不同家庭之间的样本差异,”弗林德斯大学学者、阿德莱德表观遗传学中心的罗奇博士说。
与此同时,丁斯代尔教授表示,从百慕大珊瑚礁水域周围的珊瑚保护粘液中收集的宏基因组——从靠近海岸到远洋——为增进对健康海洋环境中病毒的基因组了解提供了另一个机会。
丁斯代尔教授说:“更精确的宏基因组数据库和分析流程对于了解病毒圈和在气候变化和生物多样性丧失面前保护物种至关重要。”
